Biopharmazeutika: N-terminale Aminosäuresequenzierung von IgG-Antikörpern
In den vergangenen Jahren wird der Begriff „Biomedizin“ immer häufiger für neue pharmazeutische Medikamente verwendet. In diesen Biopharmazeutika werden proteinhaltige Wirkstoffe und Antikörper für die Behandelung eingesetzt, die mit Hilfe von biotechnologischen Prozessen wie genetischer Rekombination, Zellfusion und Zellkultur entwickelt und Hergestellt werden. Im Gegensatz dazu werden konventionelle Medikamente als „niedermolekulare Medikamente“ bezeichnet und durch chemische Synthese hergestellt. Während es sich bei beiden Arten um chemische Verbindungen handelt, haben Biomedikamente im Vergleich zu chemisch synthetisierten niedermolekularen Arzneimitteln charakteristischerweise ein viel höheres Molekulargewicht. Von den Top 10 aller 2015 weltweit verkauften Arzneimittel waren sieben Biomedikamente.
Biomedizin ist hochwirksam, nebenwirkungsarm und kann zur Behandlung eines breiten Spektrums von Krankheiten eingesetzt werden. Leider ist im Gegensatz zu niedermolekularen Medikamenten, die bisher zum Mainstream gehörten, die Massenproduktion von Biomedizin nicht auf die gleiche Weise möglich wie chemisch synthetisierte Produkte. Die Produktion von Biomedizin umfasst mehrere Prozesse, darunter Herstellung, Verfeinerung, Design der Darreichungsform und Lagerung. Um die Qualität von Biomedizin zu gewährleisten, müssen neben der Qualifizierung von Produkten auch Einflüsse von Rohstoffen und Herstellungsprozessen berücksichtigt werden. Dies bedeutet, dass das Management der Herstellung und Qualität von Arzneimitteln einen anderen Ansatz erfordert als bei chemisch synthetisierten Arzneimitteln. Gegenwärtig gibt es Richtlinien für die Bewertung der Qualität von Biomedizin. Diese Richtlinien verlangen, dass eine Merkmalsanalyse durchgeführt wird und eine Art der Merkmalsanalyse ist die N-terminale Aminosäuresequenzierung. Diese Analyse wird durchgeführt, um die aus der Gensequenz vermutete N-terminale Aminosäuresequenz mit der N-terminalen Aminosäuresequenz des biomedizinischen Produkts zu vergleichen und zu verifizieren. Die ideale Analysentechnik für diese Aufgabe ist die Aminosäuresequenzierung über die Edman-Methode. Bei dieser Technik wird die Aminosäuresequenz über den sequenziellen Abbau der Aminosäuren vom N-terminus des Proteins ermittelt. Der Proteinsequenzierer PPSQ-50A von Shimdazu ist ein System, dass diese Analysenschritte automatisiert durchführt. Dieses System erleichtert deutlich die Identifizierung von Aminosäuresequenzen vom N-Terminus der Zielproteine und -peptide.
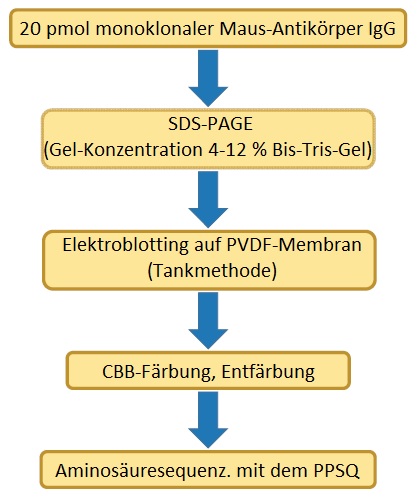
In dieser Applikation wird ein Beispiel für die Aminosäuresequenzierung des Maus-Antikörpers IgG unter Verwendung PPSQ-53A mit Gradiententrennung als Beispiel für die N-terminale Aminosäuresequenzierung von Biomedizinprodukten vorgestellt.
Methode
Die Grundstruktur der Antikörper besteht aus zwei H-Ketten (schwere Ketten mit höherem Molekulargewicht) und zwei L-Ketten (leichte Ketten mit niedrigerem Molekulargewicht).
Da intakte Antikörper ein hohes Molekulargewicht haben (ca. 150 kDa), erweist sich die Durchführung der N-terminalen Aminosäuresequenzierung mit dem Protein-Sequencer in diesem Zustand als sehr schwierig. In diesem Analysebeispiel wurden 20 pmol des monoklonalen Maus-Antikörpers IgG durch Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese (SDS-PAGE) in H-Ketten und L-Ketten getrennt und diese Ketten dann auf eine PVDF-Membran elektrogeblottet. Die PVDF-Membranen von CBB-gefärbten L-Ketten und H-Ketten wurden mit dem PPSQ-53A analysiert.
Ergebnisse der N-terminalen Aminosäuresequenzierung
Abb. 3 und Abb. 4 zeigen die Ergebnisse der N-terminalen Aminosäuresequenzierung von L-Ketten und H-Ketten aus dem monoklonalen Maus-Antikörper IgG. Jede Abbildung zeigt Chromatogramme von Zyklus 1 bis 5 für jede Probe (Zyklus 1 ist das Rohchromatogramm, andere sind subtrahierte Chromatogramme). In Abb. 3 identifizierte Zyklus 1 in den L-Ketten-Ergebnissen den Aminosäurerest des N-Terminus als Asparagin (Asp) und Zyklus 2 den zweiten Aminosäurerest des N-Terminus als Isoleucin (Ile). Nach Durchführung der Analyse bis zum 20. Rest wurde die Sequenz vom N-Terminus als Asp-Ile-Gln-Met-Thr-Gln-Ser-Pro-Ser-Thr- Leu-Ser-Ala-Ser-Val-Gly-Asp-Arg-Val-Thr identifiziert. Die Suche in der Datenbank ergab, dass es sich um die leichte kappa-Kette IgG1 handelt.
Ebenso zeigt Abb. 4 die Ergebnisse der H-Kette bis zum Zyklus 5. Die Sequenz vom N-Terminus wurde als Glu-Val-Gln-Leu-Gln-Glu-Ser-Gly-Pro-Glu-Leu-Leu-Val-Lys- Pro-Gly identifiziert, und eine Datenbanksuche ergab, dass es sich dabei um eine schwere Immunoglobulinkette handelt. Wie in diesem Beispiel gezeigt wird, ermöglicht die Durchführung der Analyse mit dem Proteinsequenzierer eine einfache und genaue Identifizierung von N-terminalen Sequenzen und zeigt, dass der Proteinsequenzierer ein wirksames System zur Steuerung der Qualität von Biopharmazeutika ist.
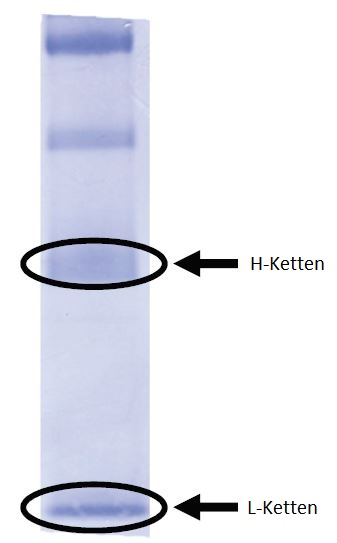
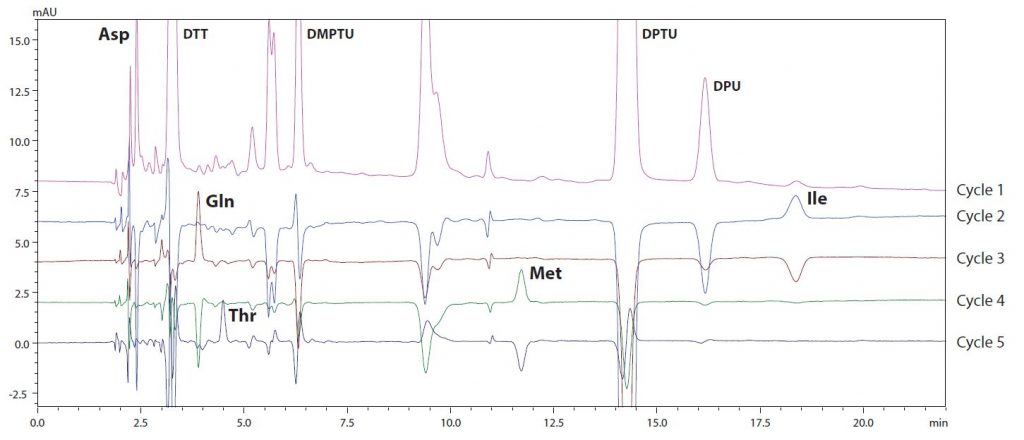
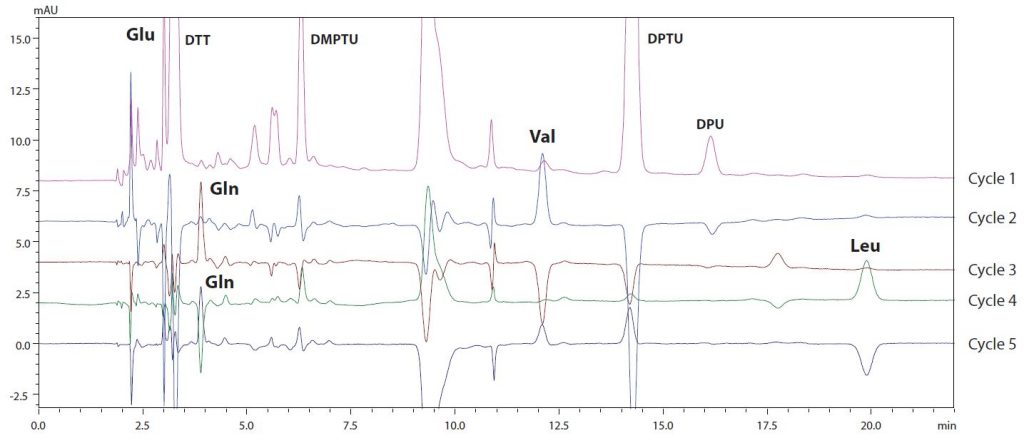
Abb. 3 (links): L-Ketten-Chromatogramme (Zyklen 1 bis 5) | Abb. 4 (rechts): H-Ketten-Chromatogramme (Zyklen 1 bis 5)