Proteinprofiling von Chemoresistenzen in Krebszellen
Im Rahmen einer Zusammenarbeit mit der Medizinischen Universität von Wien, zielte dieses Projekt darauf ab, den Nachweis von Chemoresistenzbiomarkern schneller und weniger invasiv für Patienten zu gestalten, die sich einer Chemotherapie unterziehen. Eine frühe Erkennung von Chemoresistenz verbessert die Krebstherapie und daher auch die Krebsüberlebensraten.
Krebszellen kommunizieren mit dem gesamten Organismus über extrazelluläre Vesikel (EVs), die im Körper zirkulieren und molekulare Informationen zur Unterstützung des malignen Phänotyps weitergeben.
Dickdarmkrebs-Lymphknotenmetastasen wurden mit progressiven Konzentrationen von Fluorouracil kultiviert, um eine zunehmende Chemoresistenz aufzubauen. Die EVs wurden durch Ultrazentrifugation aus dem Zellkulturüberstand entnommen (siehe Abb. 1), um als Modell für zirkulierende Krebszell-abgeleitete Biomarker-Träger aus Körperflüssigkeiten (d.h. Flüssigkeitsbiopsie) zu dienen. In dieser Arbeit wurde unter Verwendung eines MALDI-TOF MS die differentielle Expression von Proteinen in EVs als Ergebnis zunehmender Chemoresistenzen ihrer Elternzellen beobachtet (1).
Proben und Methode
Dr. Gerald Stübiger von der Medizinischen Universität Wien stellte für die Studie sowohl EVs und Zellextrakte aus Darm-Tumoren als auch gegen 5,25 und 125 µm 5-Fluoruracil (FU) resistente Lymphknotenmetastasen-Subklone zu Verfügung. Mit dem MALDI-8020 MALDI-TOF-MS-System wurden Proteinmassenspektren mit CHCA im Bereich m/z 2.000-25.000 aufgenommen und einer multivariaten Datenanalyse unterzogen (eMSTAT Solution™ Software, Shimadzu), siehe Abb. 1. Durch Verwendung von hierarchischem Clustering, PLS-DA, wurden diskriminierende Proteinmuster der EVs aus verschiedenen Zellpopulationen erhalten.

Abb. 1 Arbeitsablauf für die extrazelluläre Vesikelanalyse
Ergebnisse des Protein-Profiling
Ein repräsentatives Massenspektrum für eine der EV-Proben ist in Abb. 2 dargestellt. Die Empfindlichkeit einer solchen MALDI-MS Analytik lag im unteren Konzentrationsbereich von μg/mL (≈1.2-5 × 1010 Partikel/mL) (Daten nicht dargestellt).
Nach der Datenerfassung wurden die Peaklisten zur statistischen Auswertung in die Software eMSTAT Solution importiert. Die Replikate für jeden Probentyp wurden gruppiert. Die Software macht es dem Benutzer einfach, die differenzierenden Peaks zu identifizieren. Der Score-Plot (Abb. 3) zeigt die Unterscheidung zwischen den naiven und den fluorouracil-resistenten Probengruppen. Die fluorouracil-resistenten Gruppen weisen genügend Ähnlichkeiten auf, um zusammen gruppiert zu werden, während sie gleichzeitig genügend Unterscheidungsmerkmale aufweisen, um diese Gruppen zu trennen.
Für die Charakterisierung der Chemoresistenz erwiesen sich Signale im Bereich von m/z 2.000-7.000 als am hilfreichsten.
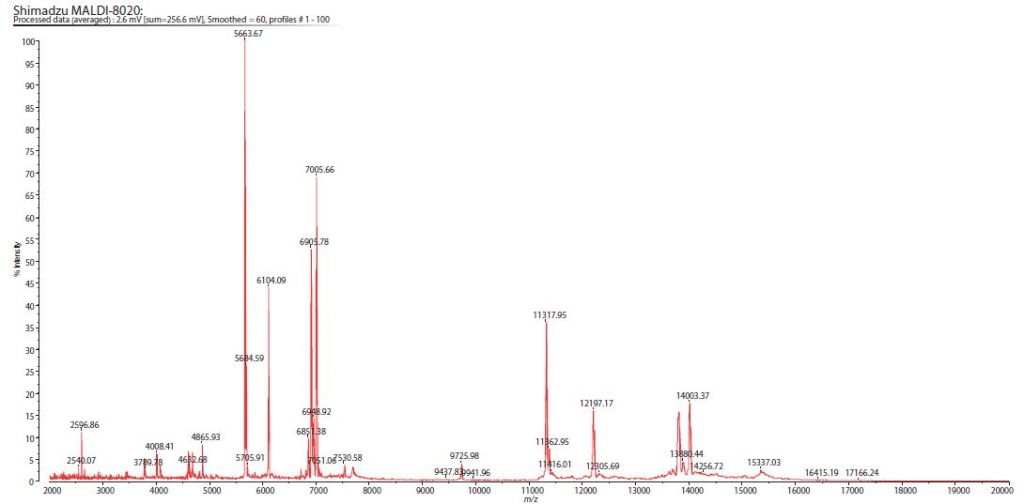
Abb. 2 Spektrum der Expression des extrazellulären Vesikelproteins aus dem primären Dickdarmkrebs
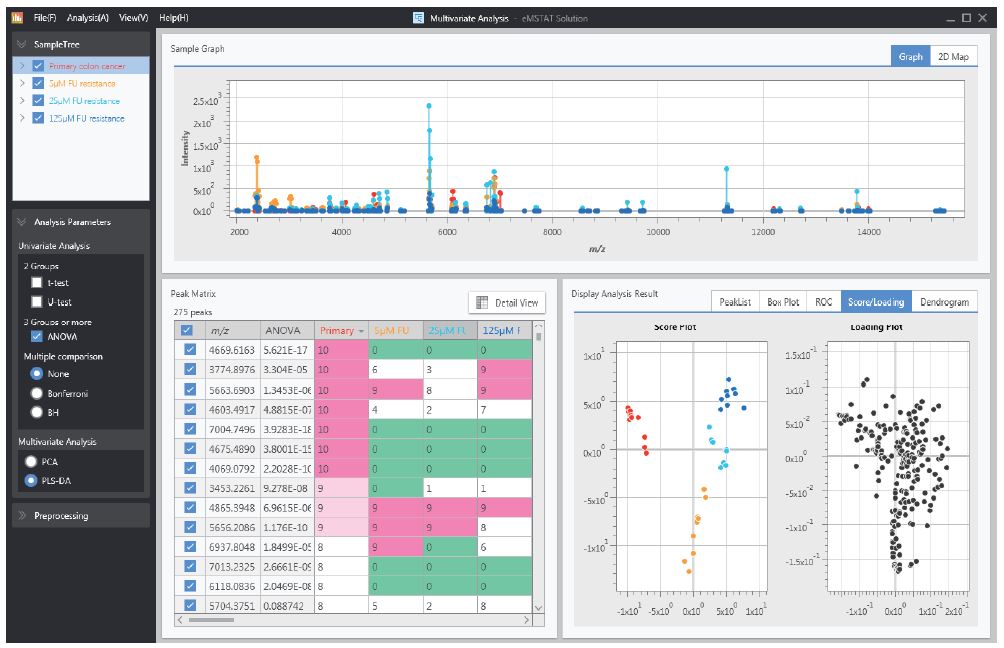
Abb. 3 PLS-DA Analyse der 4 Vesikel-Expressionsgruppen
(rot) Primärer Darmkrebs (gelb) 5 μM FU-Widerstand (blau) 25 μM FU-Widerstand (grün) 125 μM FU-Widerstand
Fazit
Diese Arbeit unterstreicht den Nutzen des MALDI-8020 in Kombination mit der Software eMSTAT Solution zur Erleichterung der Biomarker-Entdeckung.
Der MALDI-MS-Protein-Profiling-Ansatz zeigt das Potenzial, in Zukunft als neuartiges Analysesysteme für die minimal-invasive Krebsdiagnostik und Chemotherapie-Überwachung zu dienen, z.B. für die Früherkennung von Therapieresistenz ohne Biopsie.
Referenzen
(1) Stübiger, G. et al., MALDI-MS Protein Profiling of Chemoresistance in Extracellular Vesicles of Cancer Cells, Analytical Chemistry, 90, 13178-13182 (2018)