Differenzierung und Quantifizierung von Tierhaaren mit einem Benchtop-MALDI-TOF-Massenspektrometer
Matrix-unterstützte Laser-Desorptions-/Ionisations-Flugzeit-Massenspektrometer (MALDI-TOF MS) ermöglichen die einfache und schnelle Erfassung von Molekulargewichten aus einer Vielzahl von makromolekularen Proben, einschließlich Peptiden und Proteinen. MALDI-TOF MS werden häufig zur Bestimmung der Molekulargewichte von synthetischen Produkten und Naturstoffen sowohl in Forschungs- und Entwicklungslabors als auch vor Ort in der Qualitätskontrolle eingesetzt.
Eine der neuen Anwendungen von MALDI-Massenspektrometrie ist eine Methode zur Differenzierung von Tierhaaren durch den Nachweis artspezifischer Peptide in Tierhaaren 1). Diese Differenzierungsmethode ist von der Internationalen Organisation für Normung (ISO) als „Methode zur Proteomanalyse von Kaschmir und einigen anderen Tierhaarfasern“ anerkannt und durch eine ISO-Norm definiert 2). Es ist zu erwarten, dass diese Differenzierung nicht nur bei gefälschtem Kaschmir, sondern auch bei der Kontamination von Lebensmitteln usw. durch Einschluss von Fremdstoffen angewendet werden kann, da die Analyse auch mit nur einem einzigen Haar möglich ist. Dieser Artikel befasst sich mit der Differenzierung von Tierhaaren und zeigt zusätzlich ein Beispiel zur Quantifizierung artspezifischer Peptide in Tierhaaren unter Verwendung des MALDI-8020 Benchtop MALDI-TOF MS.

Probenvorbereitung
Die unbehandelten Naturfasern aus Kaschmir, Yak-, Schaf- und Alpakawolle sowie Kamel-, Pferde- und Hasenhaare wurden mit einer Schere oder einer Kugelmühle fein zerkleinert. Ein Volumen von 0,5 mL 4 %igem Natriumdodecylsulfat (SDS), welches 50 mM Dithiothreitol (DTT) in 0,1 M Phosphatpuffer (pH 7,8) enthielt, wurde 10 bis 50 mg des zerkleinerten Rohhaares hinzugefügt. Anschließend wurde diese Probe 15 Minuten bis 1 Stunde bei 95 °C erhitzt. Nach dem Erhitzen wurde dem Extrakt Iodacetamid hinzugegeben, um eine Konzentration von 100 mM zu erreichen. Das Belassen des Extraktes bei Raumtemperatur für 15 min ermöglichte die gewünschte Reaktion. Die Zugabe von 10 μL von 25 mM DTT stoppte die Reaktion.
Probenvorbereitung zur Differenzierung
Der Extrakt wurde mit Trichloressigsäure gefällt, aufgereinigt und dann erneut in einer 7 M Harnstoff- und 2 M Thioharnstofflösung aufgenommen. Der wieder aufgelöste Extrakt wurde mit 50 mM Ammoniumhydrogencarbonat verdünnt und dem Extrakt daraufhin 2,0 μg Trypsin hinzugefügt. Die Probe wurde über Nacht bei 37 °C aufgeschlossen.
Probenvorbereitung zur Quantifizierung
Mithilfe der SDS-Polyacrylamid-Gelelektrophorese (SDS-PAGE) konnte der Extrakt aufgetrennt werden. Folgend der Färbung mittels Coomassie Brilliant Blue (CBB) wurden die Banden, die das Zielprotein enthielten, aus dem Gel geschnitten. Daraufhin wurden die ausgewählten Proben durch die Verwendung von 50 mM Ammoniumhydrogencarbonat/50 % Acetonitril entfärbt und schließlich unter Zugabe von etwa 150 ng Trypsin für 1 h bei 50 °C aufgeschlossen.
Die Entsalzung der Proben konnte durch die Verwendung von ZipTip® μC18 (Merck Millipore) realisiert werden. Nach der Entsalzung wurde die Probenlösung auf das MALDI-Target aufgebracht, 0,5 μL Matrixlösung hinzugefügt und schließlich die massenspektrometrische Analyse durchgeführt. Die Matrixlösung wurde durch Auflösen von CHCA (α-Cyano-4-hydroxyzimtsäure) in 50 % Acetonitril/0,05 % Trifluoressigsäure (TFA) in einer Konzentration von 5 mg/mL hergestellt.
Für die MALDI-MS Analyse wurde das MALDI-8020, ein lineares Benchtop MALDI-TOF MS (Abb. 1), im positiven Messmodus verwendet. Die von jedem Massenspektrum erhaltene Peakliste wurde zur Diffenrenzierung mittels der statistischen Auswertesoftware eMSTAT Solution™ analysiert, um die einzelnen Tierhaare bzw. Naturfasern zu identifizieren.
Ergebnisse
| Art des Organismus | Keratin Typ I-Protein | Aminosäuresequenz | [M+H]+ Durchschnittliche Masse * |
|---|---|---|---|
| Kaschmir | Keratin 33A [Capra hircus] | YSCQLNQVQSLIVNVESQLAEIR | 2692,38 |
| Yak | Keratin Typ I Mikrofibrillar, 47,6 kDa-ähnlich [Bos mutus] | YSSQLAQVQGLIGNVESQLAEIR | 2504,81 |
| Schaf | Keratin 33B [Ovis aries] | YSCQLSQVQSLIVNVESQLAEIR | 2666,03 |
| Kamel/Alpaka | Keratin, Typ , mikrofibrillär, 47,6 kDa [Camelus dromedaries] | YGSQLSQVQGLITNVEHQLAEIR | 2584,90 |
| Pferd | Keratin 33A [Equus caballus] | YSSQLSQVQGLITNVESQLAEIR | 2564,86 |
| Hase | Keratin, Typ , Kutikular Ha3-I [Oryctolagus cuniculus] | YSSQLSQVQCMISNVESQLGEIR | 2657,99 |
| Hund | Keratin 33A [Canis lupus familiaris] | YSSQLNQVQCMITNVESQLAEIR | 2713,07 |
| Braune Ratte | Keratin 31 [Rattus norvegicus] | YSSQLSQVQCLITNVESQLGEIR | 2653,98 |
| Mensch | KRT34 protein [Homo sapiens] | YSSQLSQVQSLITNVESQLAEIR | 2594,89 |
* Die durchschnittliche Masse der Cysteinreste wird nach ihrer Carbamidomethylierung mit Iodacetamid berechnet.
Tabelle 1: Aminosäuresequenz der tierartspezifischen Region bei Keratin Typ I
Differenzierung der Tierhaare
Abb. 2 zeigt die Massenspektren von Trypsin-verdauten Peptiden aus Tierhaaren. Jedes art-spezifische Peptidsignal im Bereich von m/z 2450 bis 2750 konnte nachgewiesen werden. Durch Verwendung dieser spezifischen Signale ist es möglich, die unterschiedlichen Tierhaare nach ihrer Artenherkunft zu differenzieren.
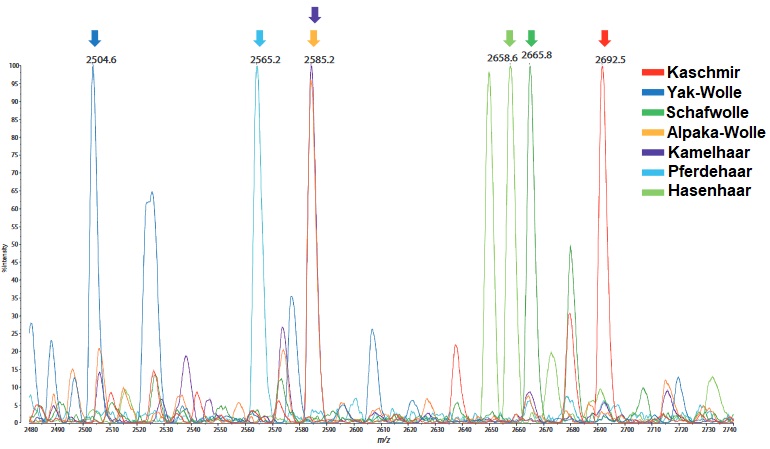
Abb. 2 Massenspektren von Trypsin-verdauten Peptiden aus Tierhaaren
Die Ergebnisse der Zuweisung von tierartspezifischen Signalen sind in Tabelle 1 1) dargestellt. Der Hauptbestandteil von Tierhaaren besteht aus dem Proetin Keratin. Diese hier ermittelten Aminosäuresequenzen stammen ebenfalls aus Keratin und sind vom Keratintyp I abgeleitet. Es ist zu erkennen, dass diese Sequenzen sowohl für Tierarten als auch für Menschen und braune Ratten spezifisch sind. Allerdings ist zu beachten, dass die Massen der spezifischen Signale für Alpaka und Kamel zusammenfallen, wodurch diese beiden Spezies mit dieser Methode nicht voneinander unterschieden werden können.
Schließlich erstellten wir unter Verwendung der in Abb. 2 gezeigten tierartspezifischen Signale und der eMSTAT-Solution eine Bestimmung der passenden Tierart (Abb. 3 links). Mit dieser Diskriminanzanalyse konnten wir die richtige Tierart von drei getrennt analysierten Proben – aus Kaschmir-, Yak- und Schafwolle – in allen Fällen bestimmen (Abb. 3 rechts).
Quantifizierung artspezifischer Peptide aus Tierhaaren
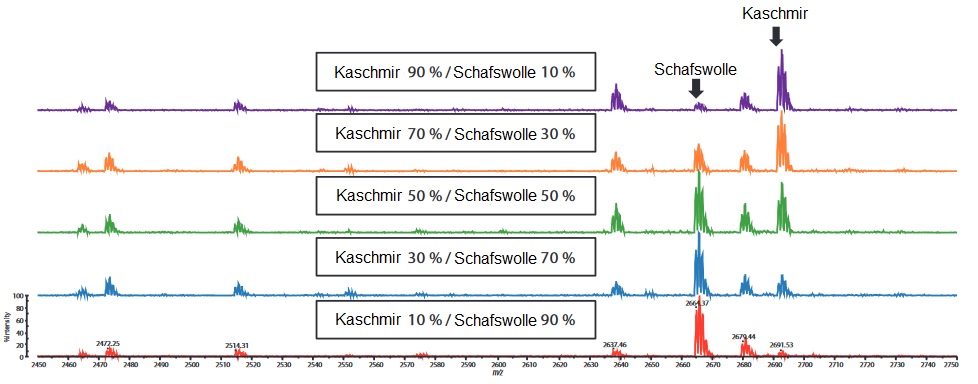
Die Abbildung 3 zeigt die Massenspektren von Trypsin-verdauten Peptiden aus Proben von Kaschmir gemischt mit Schafwolle in verschiedenen Verhältnissen von 10 % auf 90 %. Auch hier konnten die kaschmir- und wollspezifischen Peptide, abgeleitet von dem Keratin, detektiert werden. Anhand von Änderungen entsprechend dieser Tierfasermischungen in den Intensitätsverhältnissen der unterschiedlichen Signale, können sowohl die Art der Tierhaare identifiziert als auch die Mischungsverhältnisse selbst aufgenommen werden.
Im nächsten Schritt wurden die Verhältnisse der Signalintensitäten mittels folgender Formel ermittelt:
Kashmir Peak (%) = PAc * 100/ (PAc + PAw)
PAc: Intensität des kashmir-spezifischen Signals
PAw: Intensität des schafswoll-spezifischen Signals
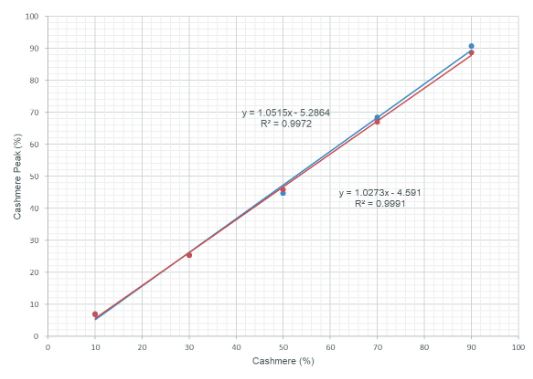
Abbildung 4 zeigt die dazugehörige Kalibrierkurve, in der das Verhältnis der erhaltenen Intensitätsverhältnisse gegen die Mischungsverhältnisse aufgetragen wurden. Um die Reproduzierbarkeit zu bestätigen, wurden die gleichen Proben der SDS-PAGE in zwei Serien durchgeführt. Für beide Serien ist eine Kalibrierkurve (rote und blaue Linie, Abb. 4) erstellt worden. Dabei ist die Linearität der Kalibrierkurve zufriedenstellend und der Korrelationskoeffizient liegt nahe bei 1.
Fazit
Es wurde gezeigt, dass Tierhaare mit dem Benchtop MALDI-8020 MALDI-TOF MS und der statistischen Analysesoftware eMSTAT Solution schnell und einfach differenziert werden konnten. Desweitere konnte die Möglichkeit der quantitativen Analyse bei Mischungsverhältnissen von Tierhaarfasern in kommerziell erhältlichen Faserprodukten aus Kaschmir, Schafwolle und Yak als Rohmaterial dargestellt werden. Es ist in Zukunft zu erwarten, dass durch eine Weiterentwicklung dieser Methode an dem MALDI-8020 sogar weitere verschiedene Differenzierungen sowie Quantifizierungen durchführbar sind, die über Kaschmir hinaus auch Federn, menschliches Haar und weitere Tierhaarfaser-Produkte umfassen.
Danksagung
Wir sind Professor Shinichi Ohashi vom Genome Biotechnology Laboratory am Kanazawa Institute of Technology für seine Unterstützung bei der Datenerfassung und der Bereitstellung von Proben zu Dank verpflichtet.
<Verweise>
1) Shinichi Ohashi et al: Quantitative analysis of cashmere and other animal hair fibers in textiles using MALDI-TOF mass spectrometry, SEN’I GAKKAISHI, 70, 6, 114-120 (2014)
2) ISO 20418-2: 2018 Textiles — Qualitative and quantitative proteomic analysis of some animal hair fibers — Part 2: Peptide detection using MALDI-TOF MS